第一本《DNA折纸初学者指南》
2006年加州理工学院首创DNA折纸技术,将长链DNA反复折叠以构建各种微小的3D结构,包括微型生物传感器和药物递送容器。过去十年,DNA折纸技术吸引了数百名新的研究人员,他们渴望建立能够检测和治疗人体疾病的容器和传感器,评估污染物对环境的影响,并协助许多其他生物应用。
虽然DNA折纸的原理很简单,但设计新结构的工具和方法并不容易掌握,也没有很好的文献记载。此外,对于构建DNA结构的最有效方法,以及如何避免可能浪费数月甚至数年研究的陷阱,新采用这种方法的科学家们还没有一个统一的参考资料。
这就是为什么美国国家标准与技术研究所(NIST)的研究人员Jacob Majikes和Alex Liddle多年来一直致力于编写第一本关于这种技术的详细教程。他们的报告提供了一个逐步指导设计DNA折纸纳米结构,使用最先进的工具。Majikes和Liddle在2021年1月8日出版的国家标准与技术研究院研究期刊(Journal of Research of the National Institute of Standards and Technology)上描述了他们的工作。
“论文会告诉每个人作者们都做了什么,但不会告诉你都经历了什么,做了哪些准备,我们想把所有人开发的工具放在一块,并解释那些你在传统期刊文献中没看到的事情,”Majikes说。
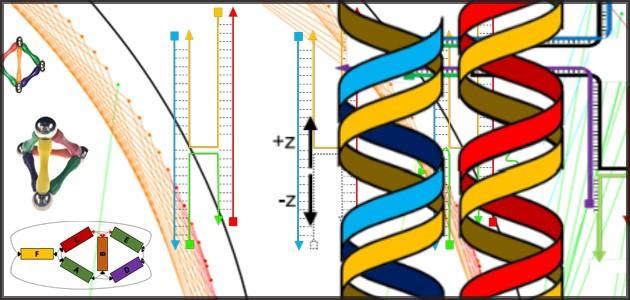
DNA折纸依赖于DNA分子互补碱基对相互结合的能力。在DNA的四个碱基——腺嘌呤(A)、胞嘧啶(C)、鸟嘌呤(G)和胸腺嘧啶(T)中,A与T结合,G与C结合。这意味着As、Ts、Cs和Gs的特定序列将找到并与补体结合。
这种结合使短链DNA起到“订书钉”的作用,用于使长链部分折叠或连接单独的链。一个典型的折纸设计可能需要250个订书钉。通过这种方式,DNA可以自组装成各种形状,形成一个纳米尺度的框架,各种各样的纳米颗粒可以附着在这个框架上,这些纳米颗粒在医疗、生物研究和环境监测方面都很有用。
Majikes说,使用DNA折纸的挑战是双重的。首先,研究人员正在用一门外语——碱基对A、G、T和C——制造3D结构。此外,他们还得利用这些碱基对订书钉扭曲和解开熟悉的DNA分子双螺旋,使链弯曲成特定形状。这可能很难设计和可视化。Majikes和Liddle敦促研究人员在开始制作之前,通过建立3D模型来增强他们的设计直觉,比如用条形磁铁制作的雕塑。这些模型可以揭示折叠过程的哪些方面是关键的,哪些方面不那么重要,然后应该“展平”为2D,以便与DNA折纸的计算机辅助设计工具兼容,因为后者通常是2D输入输出的。
Majikes指出,DNA折叠可以通过多种方式完成,有些方式效率较低。事实上,有些策略可能注定要失败。
“我们会写‘你可以这样做,但这不是一个好主意’,这种观点不可能出现在传统的期刊文章中,但因为NIST专注于推动国家的技术水平,我们能够在NIST期刊上发表这项工作,”Majikes说。“我不认为其他任何地方会给我们回旋的余地,花费人力和时间来做这些整合。”
Liddle和Majikes计划继续他们的工作,并提供一些额外的手稿,详细说明如何成功地用DNA制造纳米级设备。
原文检索:DNA Origami Design: A How-To Tutorial; https://nvlpubs.nist.gov/nistpubs/jres/126/jres.126.001.pdf
(生物通:伍松)