【Gut】成像质谱流式助力浙大一院肝胆胰团队肝细胞癌肿瘤微环境拓扑分析
肝癌是全球癌症相关死亡的第三大原因,肝细胞癌 (HCC) 是最常见的肝脏原发性恶性肿瘤。酪氨酸激酶抑制剂 (TKI) 治疗疗效仍然有限,免疫检查点抑制剂 (ICIs),如程序性细胞死亡蛋白-1 (pD-1) 阻断抗体,显示出比 TKI 更高的客观反应率和更少的总体不良副作用。然而与黑色素瘤或淋巴瘤相比,HCC 的ICI 抗肿瘤活性仅达到中等的客观反应率(15%–20%)。
由于HCC肿瘤微环境 (TME) 是高度复杂的,具有组成各种功能单元、细胞邻域 (CN) 的多种细胞成分,传统的免疫组化或免疫荧光的方法难以满足多参数检测的需求,从而限制了其在复杂TME研究中的应用。成像质谱流式技术(Imaging Mass Cytometry,简称IMC),利用数十种稀有金属标记抗体,对单个组织切片进行染色,可以同时检测数十种蛋白质和蛋白质修饰,并保留细胞空间信息,确定空间分辨的单细胞表型,即能够实现基于空间分辨率的 HCC TME 表型是TME 拓扑分析的实用工具。
近日,来自浙江大学梁廷波教授、盛剑鹏研究员带领的研究团队在HCC免疫治疗领域获得了突破进展。他们从大量亚细胞空间分辨率的蛋白数据中,首次揭示了 HCC TME 的各种拓扑功能单元,同时提供了最大的 HCC 病理景观库,突出了Kupffer 细胞特异性靶向而不是整体骨髓细胞阻断作为 HCC 治疗的新型免疫疗法的潜力。研究结果发表在Gut(IF=23.06)期刊。
这项研究利用IMC量化了134名 HCC 患者和7名健康供体的 FFpE切片样本中 36 个 marker 的表达和定位信息,获得了562张亚细胞空间分辨率的高维肿瘤病理图像。图像中包含人肝脏微环境区域独特的组织结构,以及主要基质细胞类型和免疫细胞群(Fig 1)。
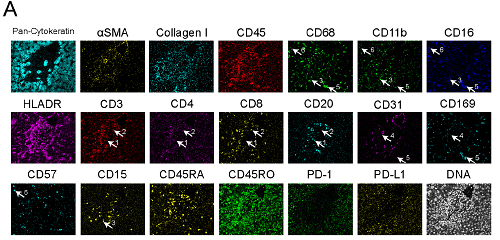
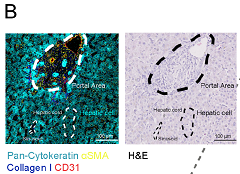
Fig1.IMC获得正常肝组织包含36个marker的表达代表性数据的高维图像。(A)marker单色伪彩图片,箭头1: B细胞(CD3- CD20+),箭头2: CD8+ T细胞(CD3+ CD8+ CD4-),箭头3: 中性粒细胞(CD11b+ CD68- CD15+),箭头4:浸润性巨噬细胞(CD11b+ CD68+ CD16- CD169low),箭头5: kuffer细胞(CD11blow CD68+ CD16+ CD169+),箭头6: 自然杀伤(NK)细胞(CD68- CD16+ CD57 +);(B) 描绘肝脏组织结构,左图pan-keratin (青色)、αSMA(黄色)、collagen I (蓝色)和CD31(红色),肝细胞呈青色,肝索、肝窦用白色虚线突出;右图H&E染色,黑色虚线突出相同结构,比例尺100 μm。
如何将图像信息解析出来并与临床数据建立联系,成了下—步研究的关键。研究人员对高维数据进行深入挖掘。
HCC微环境的组织拓扑分析
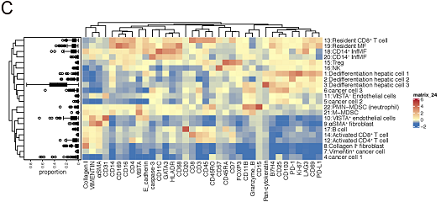
对于组织拓扑分析,研究人员首先利用Cellprofiler软件分割出组织图像中的每个细胞,从而得到单细胞水平的marker表达数据。将分割数据输入hisoCAT软件进行tSNE和phenoGraph和细胞邻域(CN)等分析。将组织细胞划分成了22 个元簇(metacluster),包括3个肝细胞元簇、4个癌细胞元簇、2个内皮细胞元簇、2个成纤维细胞元簇和11个免疫细胞元簇。热图展示每个元簇中marker的表达,发现许多非免疫细胞表达pD-1,且去分化的肝细胞1和2表达的pD-1最高(Fig 2)。
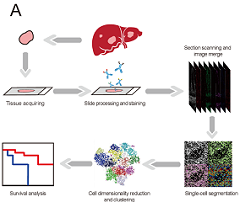
Fig 2. IMC数据处理和分析流程。图像单细胞化后,根据单细胞表达数据聚成的22个元簇 (A) IMC数据采集示意图,包括组织制备、抗体染色、图像采集与组装、单细胞识别、细胞降维、聚类和生存分析; (C)热图显示元簇marker的平均表达,右侧箱形图显示其在不同患者中的频率分布模式。
HCC三种主要TME类型中免疫细胞和基质细胞特异性分布
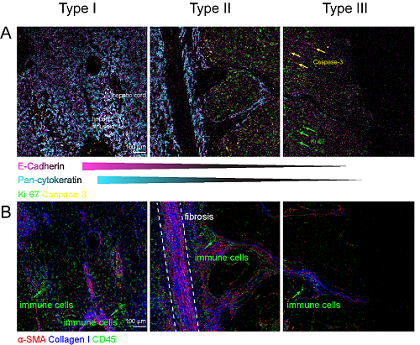
分析了每个肝癌患者样本中的多个区域,在HCC中发现了三种主要类型的TME(I-III型),具有不同的基质和免疫细胞分布模式,这些模式对应于肝细胞去分化的不同阶段。I型:肝细胞标记物, 如pan-keratin和E-cadherin强表达,肝脏的主要结构,包括肝窦和肝索保存完整,这表明I型区域类似于正常肝组织和癌旁组织;II型:成纤维细胞标记物,如αSMA和collagen I在区域高度富集,反映纤维化主要发生在该区域;III型:肝细胞失去了上皮标记物的表达,如pan-keratin和E-cadherin,提示肝细胞发生去分化,且增殖标记物Ki-67在III型区域表达水平高于其他区域(Fig 3A)。CD45+免疫细胞主要存在于I型和II型区域,而III型区域几乎没有这些细胞(Fig 3B)。这些结果揭示了三种主要类型TME和瘤内区域,表现出肝细胞去分化,间质细胞和免疫细胞的区域特异性分布。
Fig 3.三种主要类型肿瘤内区域的IMC图像。(A)E-cadherin (红色), pan-keratin (青色)、Ki-67(绿色)和caspase-3(黄色) 的叠加图片,用于识别HCC的I型、II型和III型区域。黄色和绿色箭头分别表示Caspase-3和Ki-67染色。(B)αSMA(红色)、collagen I (蓝色)和CD45(绿色)的叠加图观察纤维化区域和免疫细胞分布模式。绿色箭头表示免疫细胞。白色虚线突出纤维化区域。
单细胞分析不同TME中细胞的差异分布
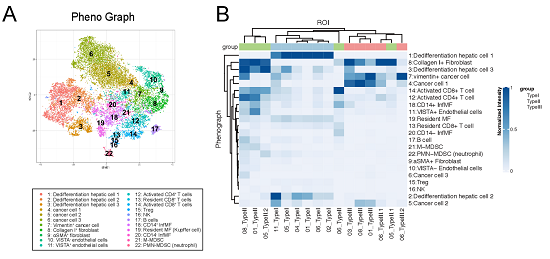
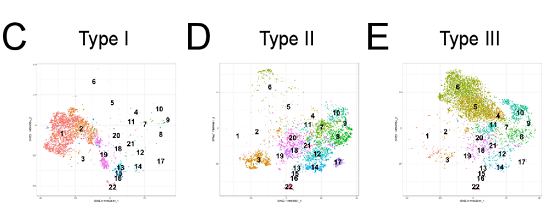
作者进一步研究去分化过程与肿瘤微环境变化之间的关系,采用tSNE在单细胞水平上分析,并在tSNE图中添加元簇(1-22)注释(Fig 4A)。热图分析三种HCC TME中元簇频率 (Fig 4B)。肝细胞、成纤维细胞、内皮细胞和不同的免疫细胞群分离成不同的元簇。根据pan-keratin和E-cadherin的表达减少,在I型区域有2个肝细胞元簇(1-2)。元簇1细胞显示最强的pan-keratin和E-cadherin染色,而元簇2的肝细胞pan-keratin和E-cadherin表达减少,提示I型区去分化过程开始(Fig 4C)。II型区域的元簇3去分化肝细胞保持正常水平的pan-keratin,而E-cadherin表达有限(Fig 4D)。在III型区域有三群癌细胞(元簇4-6),缺乏pan-keratin和E-cadherin染色,代表大部分去分化细胞 (Fig 4E)。因此,对高维IMC图像的单细胞分析详细地证实了细胞在I-III型区域的差异分布。
Fig 4. 不同区域的细胞组成和功能变化。 (A)tSNE展示HCC TME的元簇组成;(B) 不同区域22个元簇频率分布热图; (C-E)分别为I-III型区域tSNE图
肝癌微环境内细胞邻域与患者生存的相关性
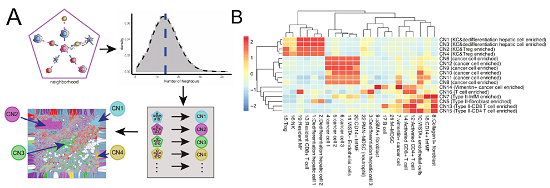
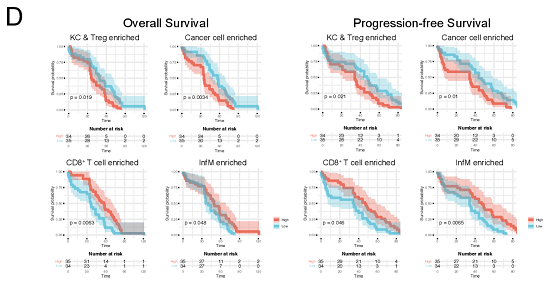
我们生活的城市由不同的社区组成(如工业、住宅或农业),社区是城市特定功能发生的区域,如工业产出或能源消耗。在本研究中,TME相当于城市,而CN 相当于城市中的社区。作者将CN定义为中心细胞4 μm内的初级邻近细胞及初级邻近细胞的次级邻近细胞(Fig 5A)。根据CN内的主要细胞簇对CNs进行注释,分成了16个CN (Fig 5B)。为了确定CN功能单位是否与患者预后相关,作者对拓扑功能单元与患者总生存期(OS)和无进展生存期(pFS)之间关系进行探索。结合69例未接受新辅助治疗或肝移植的患者的Kaplan-Meier生存分析,生成每个患者的拓扑图,计算CN功能单位频率。发现癌细胞富集的CN (CN9)与患者OS和pFS较差相关,CD8+ T细胞富集的CN (CN13)与患者OS延长有关。且KC-富集的CN、InfM-CN与患者预后呈相反关系。高频率KC富集的CN (CN4)与患者OS和pFS较差相关,而InfM-CN (CN7)较高频率与患者OS无显著相关(Fig 5D)。这些结果表明,肝癌的拓扑功能单位可能成为新的肝癌患者预后生物标志物。此外, KC和InfM组成功能相反的CNs肝癌TME。KC-CN频率高的患者预后较差,而infM-CN频率高的患者预后较好。
Fig 5. 肝细胞癌肿瘤微环境细胞功能单位与患者预后的相关性。(A)CN分析方法;(B) 13种主要的CN类型首先通过CN内的主要细胞类型进行注释,然后通过CN中每个细胞类型的缩放频率(如图y轴所示)进行聚类; (D)按照A所示的程序对患者的IMC图像进行拓扑分析,然后计算每个患者的CN频率。比较不同类型CN频率的高或低与患者的总生存期和无进展生存期的关系。
Kupffer细胞的耗尽增强肿瘤抗pD-1治疗的反应
接下来作者构建小鼠模型,利用IMC等技术,证明了肝脏中Kupffer细胞的消耗在很大程度上增强了T细胞反应,减少了肝脏肿瘤的生长并且肿瘤对抗pD-1治疗反应敏感。突出了Kupffer 细胞特异性靶向而不是整体骨髓细胞阻断作为 HCC 治疗的新型免疫疗法的潜力。
这项研究成功地采用Fl uidig m的组织成像质谱流式系统对肝细胞癌肿瘤免疫微环境进行深入的拓扑分析。与传统的免疫组化或者免疫荧光不同,IMC采用金属元素作为抗体的标签,利用质谱系统进行信号的检测。从原理上避免了荧光串色、组织背景等因素对结果的影响,可在一次扫描中同时检测几十种蛋白质,已成为肿瘤微环境拓扑分析的有效工具,是研究健康组织或病理组织的理想手段。
参考文献:
1 Sheng, J.p., Zhang, J.L., Wang L. et al. " Topological analysis of hepatocellular carcinoma tumour microenvironment based on imaging mass cytometry reveals cellular neighbourhood regulated reversely by macrophages with different ontogeny." Gut . 12 July 2021, doi: 10.1136/gutjnl-2021-324339.