Cell:癌症基因组中的结构变异比我们想象的更复杂
癌症基因组通常包含了数百个体细胞DNA重排,有些是简单的结构变异,比如缺失和易位,有些则是复杂的结构变异,如染色体碎裂。然而,有些结构变异却无法归到其中一类。
近日,威尔·康奈尔医学院和纽约基因组中心等机构的研究人员利用一种新颖的基因组分析方法,重点研究了数千个肿瘤全基因组序列,在癌症基因组中发现了一些新的复杂基因组重排。
这项成果于10月1日发表在《Cell》杂志上。文章的通讯作者、威尔·康奈尔医学院的Marcin Imielinski及其同事在文中写道:“根据基因组图谱的特征对肿瘤进行聚类,我们鉴定出与DNA修复缺陷和预后不良有关的特征。”
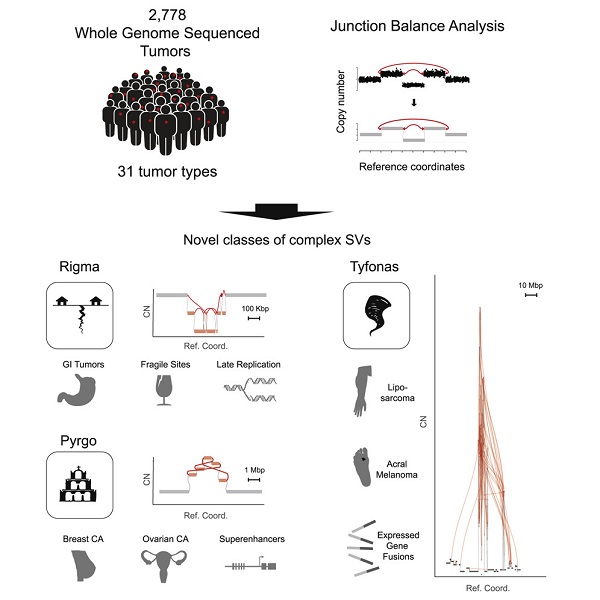
研究人员对近2,800个新的和之前报告的肿瘤全基因组序列进行分析,这些样本覆盖31种癌症类型。他们提出了一种称为连接平衡分析(JaBbA)的计算策略,试图根据肿瘤的“连接拷贝数(JCN)”图谱来搜索结构变异。
随后他们发现了三种主要类型的结构变异,分别称为“rigma”、“pyrgo”和“tyfona”重排。其中rigma是脆弱位点的缺失“裂谷”;pyrgo是与超级增强子相关的“塔形”重复;而tyfona一词则来源于“台风”,指的是扩增的折叠倒位。
研究人员表示,这些改变发生在各种类型的癌症中,影响了肿瘤基因组的大部分。Imielinski解释说:“这就像我撕下了一本书第三章的一部分,复印了五次,然后将其粘贴到第二章的中间,再把整个内容打乱,分散到这本书的最后十章中。这就是我们正在鉴定的各种改变。”
例如,他们从胃肠道肿瘤中发现了具有代表性的rigma结构变异,这些变异似乎在肿瘤演化的早期就出现了。此外,他们发现,乳腺癌和卵巢癌似乎更容易发生pyrgo改变,而tyfona变异经常出现在黑色素瘤中。
之后,他们对一个已知携带tyfona结构变异的小细胞肺癌细胞系开展后续分析。他们结合短读长序列、光学图谱分析、Hi-C三维互作图谱分析、荧光原位杂交、RNA序列以及其他数据,对这些结构改变及同时发生的突变进行更深入地观察。
研究人员表示,他们正将这种结构变异检测策略应用于癌症病例,并希望这种方法在临床和科研实验室中得到更广泛的应用。
作者总结道:“对超过10,000个癌症基因组进行大型的荟萃分析,这有助于将基因组的新颖拓扑特征与临床变量联系起来,包括治疗反应。未来,人们有望去研究急性黑色素瘤中tyfona的存在与免疫检查点抑制剂的疗效有何关系。”(生物通 薄荷)
原文检索
Distinct Classes of Complex Structural Variation Uncovered across Thousands of Cancer Genome Graphs
DOI:https://doi.org/10.1016/j.cell.2020.08.006