人类肉眼不可见的福尔摩斯:一种合成微生物
每年约约有4800万美国人因食源性病菌患病,导致约128000人住院治疗,3000人死亡。这一公共卫生问题因产品召回造成数十亿美元的经济损失而雪上加霜,凸显了迅速准确确定食源性疾病来源的必要性。
然而,随着全球供应链对消费者可获得的各种食品的日益复杂,追踪受污染物品的确切来源的任务可能会变得困难。
哈佛医学院的科学家们开发出了一种新的解决方案,可以帮助确定农产品和其他商品的原产地,这种DNA条形码微生物系统可用于以廉价、可扩展和可靠的方式标记物体。
北京时间6月5日,该研究小组在《Science》杂志上发表报告,描述了如何将合成的微生物孢子安全地引入物体和表面源发地,如田地或制造厂,并在数月后被检测和鉴定。
这些孢子来自面包酵母和一种常见的细菌菌株,广泛应用于各种用途,如益生菌膳食补充剂,并被设计成不能在野外生长,以防止不利的生态影响。
近年来,科学家们对微生物与环境之间的相互作用有了大量的了解。研究表明,家里、手机上、人体上以及其他地方的微生物群落都具有与指纹相似的独特成分。然而,尝试使用微生物指纹来鉴定出处可能会耗费时间,而且不容易成规模进行。
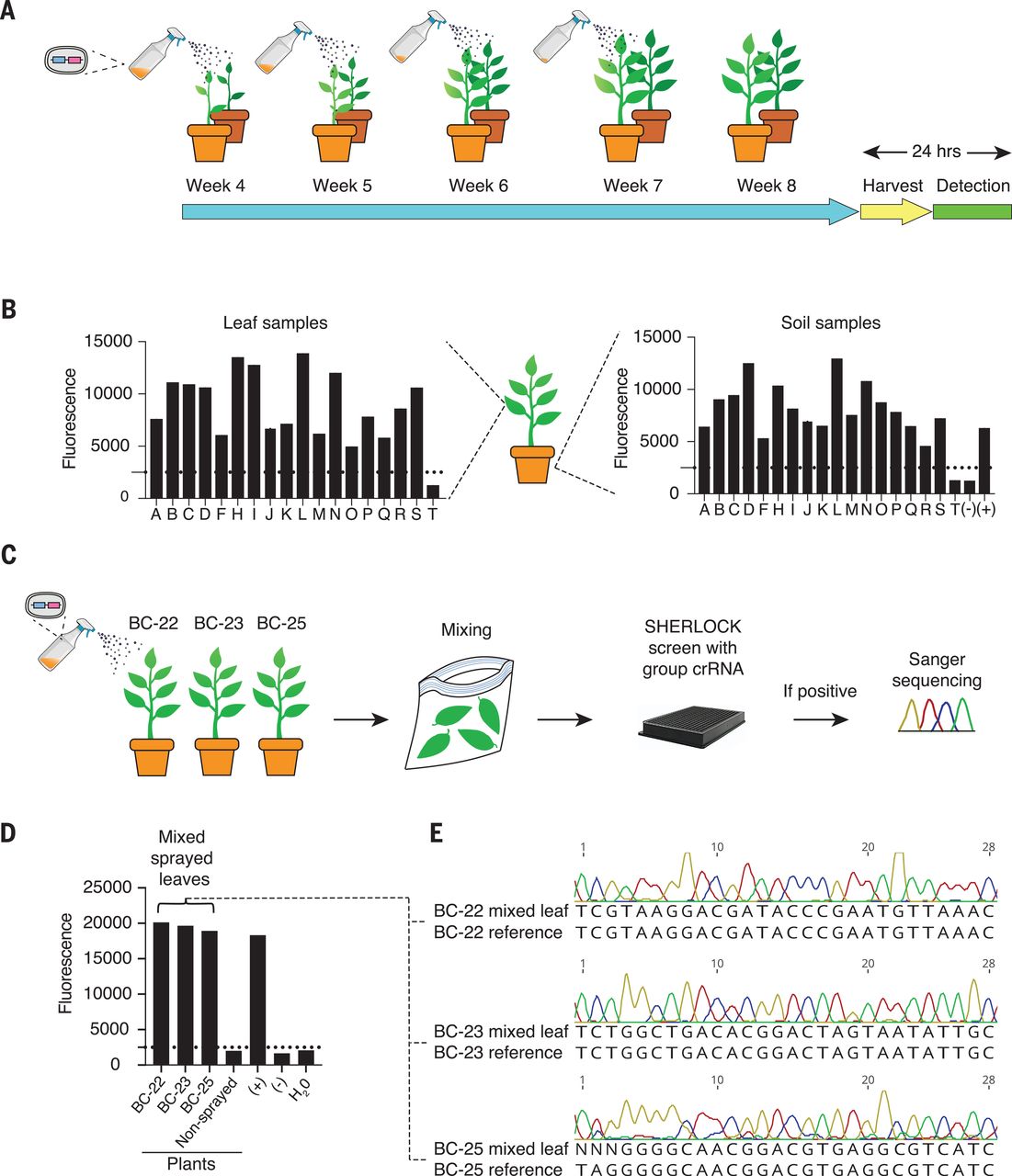
而使用定制的合成DNA序列作为条形码原则上已被证明是有效的食品标签。为了广泛应用,DNA条形码必须以低成本大量生产,在高度多变的环境中持续存在于物体上,并且能够可靠而快速地解码——这些障碍至今尚未克服,因为DNA是脆弱的。
系统生物学副教授Michael Springer 团队着手确定包装在微生物孢子中的DNA条形码是否有助于解决这些难题 ,许多微生物,包括细菌、酵母菌和藻类,在恶劣的环境条件下形成孢子。与种子类似,孢子能让微生物保持非常长的休眠期,并在高温、干旱和紫外线辐射等极端条件下生存。
研究小组创建了定制的DNA序列,将它们整合到两种微生物的孢子基因组中——酿酒酵母(也称为面包酵母)和枯草芽孢杆菌(一种常见且广泛存在的细菌,具有多种商业用途,包括作为膳食益生菌,一种土壤接种剂和某些食物中的发酵剂。这些孢子可以在实验室大量廉价地生长。
合成的DNA序列很短,不编码任何蛋白质产品,因此在生物学上是惰性的。这些序列被串联插入基因组,这样就可以产生数十亿个独特的条形码。
研究小组还确保了DNA条形码孢子不能在野外繁殖、生长和传播。他们通过使用需要特殊营养补充的微生物菌株和删除孢子萌发和生长所需的基因来做到这一点。从数亿到超过一万亿的改良孢子的实验证实了它们不能形成菌落。
为了读取DNA条形码,研究人员使用了一种廉价的基于CRISpR的工具,该工具能够快速、高灵敏度地检测到基因目标的存在。这项技术被称为夏洛克(SHERLOCK),是由麻省理工学院(MIT)和哈佛大学Broad学院(Harvard)合作开发的,由该学院成员James Collins和张峰领导。
“孢子可以在野外生存很长时间,而且它也是整合DNA条形码的好媒介,”研究的第一作者之一、英国皇家科学院系统生物学研究生詹森•钱(Jason Qian)说。“识别条形码很简单,用手机摄像头上的读板器和橙色塑料过滤器就行。我们不认为这对野外作业部署有任何挑战。”
Springer说:“李斯特菌、沙门氏菌和大肠杆菌等有害食源性病原体的爆发自然而频繁。简单、安全的合成生物学工具和基础生物学知识使我们能够创造出在解决现实世界安全问题方面具有很大潜力的东西。”
原文检索:Barcoded microbial system for high-resolution object provenance
(生物通:伍松)