Nat Commun | 何爱彬研究组揭示造血干细胞起源的表观遗..
2022年1月17日,北京大学分子医学研究所、生命科学联合中心何爱彬研究组在NatureCommunications杂志在线发表研究论文“pre-configuring chromatin architecture withhistone modifications guides hematopoietic stem cell formation in mouse embryos”,从跨尺度的染色质三维结构、组蛋白修饰及造血相关转录因子RUNX1的表观调控维度,揭示了HSC起源的命运决定机制。
造血干细胞(hematopoietic stem cell, HSC)维持整个造血系统的细胞群体组成与功能。内皮-造血转化(endothelial-to-hematopoietictransition, EHT)是HSC重要的起源过程:在背主动脉的腹侧,部分早期动脉内皮细胞(early arterialendothelial cell, eAEC)特化为生血内皮细胞(hemogenic endothelial cells, HEC),产生造血干细胞前体(pre-HSC),进而成熟发育为长期造血干细胞(long-term hematopoietic stemcell, LT-HSC)。虽然单细胞转录组分析及功能实验验证剖析了从eAEC向HSC发育的动态轨迹上的不同细胞群体1, 2,但是,HSC谱系起源和命运决定是如何受到包括染色质三维结构、组蛋白修饰及转录因子的多维表观机制整合调控的,则尚未可知。
为了探究哺乳动物胚胎中多维表观遗传层级如何调控HSC发生这一科学问题,该研究突破了少量细胞检测的技术瓶颈,应用少量细胞sisHi-C(small-scale in situ Hi-C)技术3和何爱彬团队于2019年开发的少量细胞itChIp-seq(indexing and tagmentation-based chromatinimmunoprecipitation sequencing)技术4,分别在数百个细胞中,检测染色质互作结构、组蛋白修饰及转录因子结合图谱。作者从小鼠胚胎的主动脉-性腺-中肾区和胎肝中分别收集了HSC发育路径上相邻的四种细胞类型:eAEC、HEC、pre-HSC以及LT-HSC(图1)。结合团队近期发表的单细胞转录组数据1, 2,以大范围到小范围的不同层级染色质空间结构发育变化为主线,进行HSC起源的多维表观遗传调控机制解析。
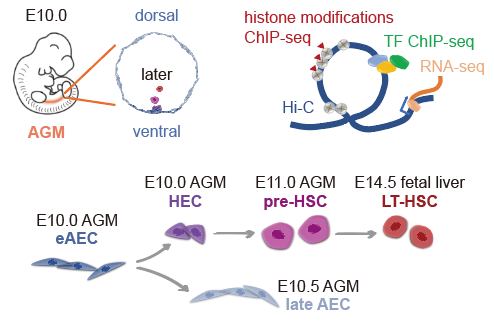
图1.细胞样品示意图及多维表观调控检测
促进造血发生的染色质互作变化发生在拓扑结构域(topologically associated domain, TAD)内部。在大尺度的染色质区室层级(100 Mbp),基因组被划分为转录活跃性高的A类区室和转录活跃性低的B类区室。仅有约10.78%的基因组区域存在任两个时期之间的A/B类区室互换的现象。TAD为染色质区室的下一级结构,其边界富集了H3K4me3信号,存在阻隔强度的动态变化,但并未直接影响TAD边界附近基因的表达。而进一步探究发现,TAD内部的调控元件互作及伴随的组蛋白修饰变化,直接与造血发育进程相关。
HSC特异的增强子在eAEC中已经处于一定程度的激活状态。与以往的从无到有的增强子激活认知不同的是,在EHT初期的eAEC中,造血过程相关TAD中的增强子已经显著富集激活性组蛋白修饰(H3K27ac和H3K4me1)。从eAEC经过HEC,到pre-HSC,增强子的活性信号并没有变化,仅在pre-HSC至LT-HSC这一阶段才进一步增强。这提示在eAEC中,已经初步准备好了造血发生的相关染色质修饰基础,但需要染色质互作结构及转录因子的结合,驱动促进HSC产生(图2)。
染色质互作在早期变化最显著。EHT初期(eAEC-HEC),TAD内部染色质互作大幅度变化,造血相关基因所在区域的互作显著增强。在EHT末期pre-HSC到LT-HSC转变中,TAD内互作只是小幅度增强,但标记活性增强子的组蛋白修饰H3K27ac则一定程度显著提升。整个过程中,相应的抑制性组蛋白修饰H3K27me3逐步减弱,为造血发生创造活跃染色质环境(图2)。
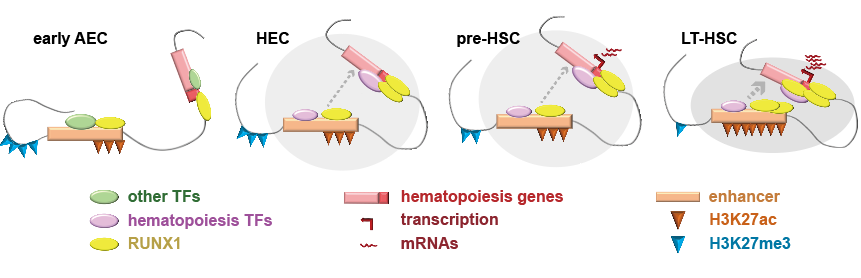
图2. HSC发生的多维表观遗传层级调控示意图
令人意外的是发现早在eAEC时期,RUNX1蛋白已富集结合于增强子-启动子(E-p)互作的锚点区域。RUNX1 是一个重要造血发育相关转录因子5,一般认为它为驱动HSC的发生所必须,其表达时间与HSC一致,它的缺失会造成胚胎造血异常或致死6, 7。受限于检测技术对细胞数目的要求,很多已有研究只能使用体外分化样品或细胞系进行RUNX1的ChIp-seq实验8, 9,10,以探究RUNX1如何调控HSC细胞命运。利用新开发的高灵敏度itChIp-seq技术,该研究以100-500个分选细胞为起始样品,检测了eAEC、HEC、pre-HSC和LT-HSC的RUNX1全基因组结合图谱。研究发现,HSC发生过程中,RUNX1参与了约40.9%的E-p互作。其中,互作强度暂时或持续上升的E-p互作与造血及免疫过程显著相关(图3)。基于RUNX1结合互作的启动子及增强子区域,该研究预测出与RUNX1协同调控染色质互作的其它转录因子,如GFI1b、pU.1、IRF家族蛋白、SMAD家族蛋白等。该预测结果为RUNX1协同其它转录因子共同调控EHT的机制探究提供了指示方向。
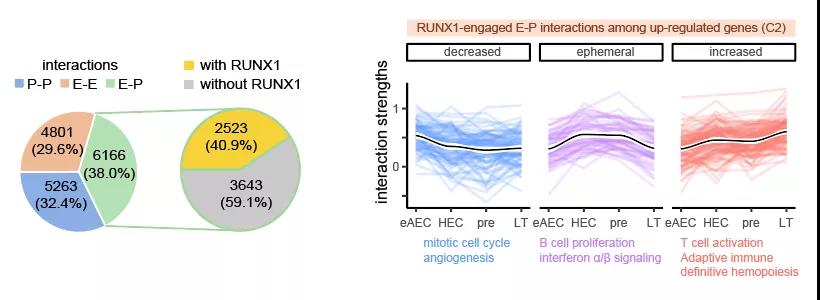
图3. RUNX1参与增强子-启动子(E-p)互作及调控相关生物学过程
简而言之,该研究突破了体内样品细胞数目限制的技术瓶颈,整合了多层级染色质结构、不同组蛋白修饰及转录因子RUNX1的多组学数据,揭示了HSC起源的表观遗传层级调控新机制。
何爱彬教授、刘兵研究员(解放军总医院第五医学中心)、兰雨研究员(暨南大学)为本文共同通讯作者。北京大学分子医学研究所博士李晨、军事科学院博士生张广雨为论文共同第一作者,感谢清华大学颉伟教授对sisHi-C技术的分享。该研究获得了科技部干细胞专项、国家自然科学基金委、广东省重点研究开发项目、生命科学联合中心、北京大学高性能计算中心和生命科学学院凤凰平台的大力支持。
原文链接:
https://www.nature.com/articles/s41467-022-28018-z
参考文献:
1. Hou S, et al. Embryonic endothelial evolution towards first hematopoietic stem cells revealed by single-cell transcriptomic and functional analyses. Cell Res 30, 376-392 (2020).
2. Zhou F, et al. Tracing haematopoietic stem cell formation at single-cell resolution. Nature 533, 487-492 (2016).
3. Du Z, et al. Allelic reprogramming of 3D chromatin architecture during early mammalian development. Nature 547, 232-235(2017).
4. AiS, et al. profiling chromatin statesusing single-cell itChIp-seq. Nat CellBiol 21, 1164-1172 (2019).
5. Chen MJ, Yokomizo T, Zeigler BM, Dzierzak E, Speck NA. Runx1 is required for the endothelial to haematopoietic cell transition but not thereafter. Nature 457, 887-891 (2009).
6. Lacaud G, et al. Runx1 is essential for hematopoietic commitment at the hemangioblast stage of development in vitro. Blood 100, 458-466 (2002).
7. YokomizoT, et al. Runx1 is involved inprimitive erythropoiesis in the mouse. Blood 111, 4075-4080 (2008).
8. Wilson NK, et al. Combinatorial transcriptional control in blood stem/progenitor cells: genome-wide analysis often major transcriptional regulators. CellStem Cell 7, 532-544 (2010).
9. Gilmour J, Assi SA, Noailles L, Lichtinger M, Obier N, Bonifer C. The Co-operation of RUNX1 with LDB1, CDK9 and BRD4 Drives Transcription Factor Complex Relocation During Haematopoietic Specification. Sci Rep 8, 10410 (2018).
10. Nottingham WT, et al. Runx1-mediatedhematopoietic stem-cell emergence is controlled by a Gata/Ets/SCL-regulated enhancer. Blood 110, 4188-4197 (2007).






























