生命学院马润林研究组在反刍类动物进化研究中取得重要进展
中国科学院大学生命科学学院马润林教授研究团队在动物主要组织相容性复合体(Major Histocompatibility Complex, MHC)结构研究中取得重要进展,初步揭示包括牛羊在内的全部反刍类动物MHC在Class II区域内存在一大段常染色体插入,把原本完整的Class II分割为IIa与IIb,并伴随有调控免疫细胞的DY类新基因产生。研究认为该染色体倒位事件很可能源自反刍类动物最后共同祖先生殖细胞发生的一次染色体倒位(Crossing Over),倒位促进了反刍类在地球生物圈的繁荣与扩张,衍生出食草类反刍动物的庞大类群,对维持自然界食物链生态平衡发挥了重要作用。该项研究结果最近发表在国际免疫学前沿杂志《Frontiers in Immunology》。
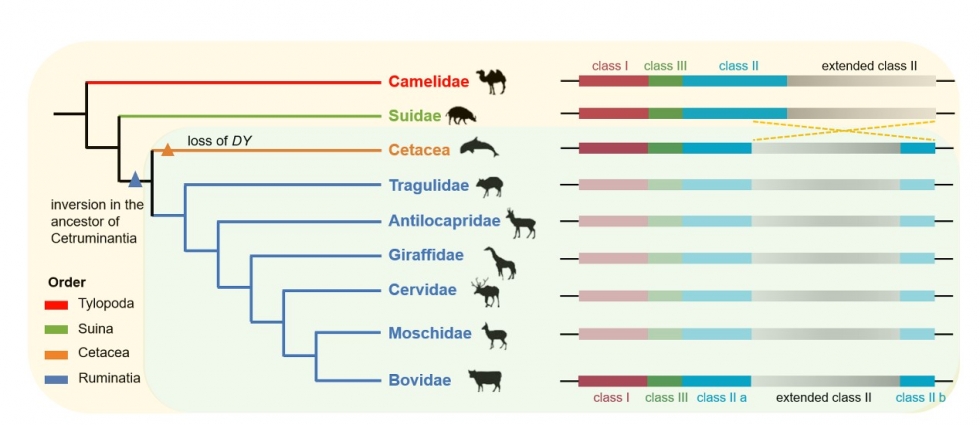
MHC编码一大类免疫防御相关蛋白与调控因子,是人类和所有哺乳动物免疫抵抗各类微生物侵染和致病性最为关键的遗传基础。反刍动物利用瘤胃微生物有效转化固定于饲草中的太阳能,对于维持地球高等肉食动物的食物链起到了承上启下的关键作用。马润林研究组在前期研究中证实家牛和绵羊MHC区域内有一段约18 Mb染色体倒位,造成MHC class II区域被分割为II a和II b两个亚区。反刍动物特有的DY基因位于倒位边缘并且在树突状细胞中高度表达。但该倒位在反刍动物中是否具有普遍性以及对反刍动物进化的意义尚属未知。
 鲸偶蹄目祖先染色体倒位的假说
鲸偶蹄目祖先染色体倒位的假说
马润林研究组使用高通量测序技术对覆盖旋角羚MHC区段的47个BAC克隆进行逐一测序,得到了全长为3,224,151bp的分子序列,注释了150个编码基因、50个tRNA基因和14个非编码RNA基因。经过序列比较分析发现旋角羚MHC的组织结构与绵羊和家牛高度保守,均有大片段染色体倒位的存在。系统发生分析表明DY基因和DQ基因的关系较近,二者的分歧时间在距今1.4亿年前。同源建模结果显示,旋角羚DY的整体结构与HLA-DQ2相似。然而,p1、p4、p6和p9这四个影响抗原肽结合的口袋的体积大小、疏水性和电荷等性质却大不相同,提示旋角羚DY和HLA-DQ2所呈递的抗原肽种类不同,理论上可以提高反刍动物对瘤胃微生物的调节能力,从而提高反刍动物对牧草的利用能力。这项研究成果加深了我们对MHC区域进化的理解,并为反刍动物共祖先染色体假说提供了额外的支持性证据,即反刍动物最后共同祖先MHC区域的古老染色体倒位可能是地球上反刍动物进化成功的原因之一。
该论文于2020年2月25日在Frontiers in Immunology杂志上在线发表。马润林研究组的博士研究生李超昆为该文章的第一作者,马润林研究员为通讯作者。相关研究得到中科院国际伙伴计划和国家自然科学基金项目的资助。






























