Nature子刊揭示动物中DNA转座子通过两种机制介导基因重复
1950年Barbara Mclintock 首次在玉米中发现转座子,并由此获得诺贝尔奖。长期以来,转座子本身被认为是垃圾DNA,但现在它们被认为是宿主基因组演化的重要推动力。转座子类型众多,包含non-LTR(Long Terminal Repeat)型逆转座子、 LTR型逆转座子、Helitron型DNA转座子、TIR(Terminal Inverted Repeat)型DNA转座子等。它们可引起包含基因重复(gene duplication)在内的各种遗传突变。实验证据表明non-LTR型逆转座子在人类基因组中介导基因重复,产生大量新基因[1];中国科学院动物研究所张勇研究组于2016年发现动物中,LTR可借由模板跳转介导基因重复[2];Helitron在蝙蝠中通过通读(transduction)的形式介导基因重复[3]。到目前为止,TIR型转座子介导基因重复的能力只在植物中被揭示,其中水稻中的MULE元件捕获大量宿主基因,形成pack-MULE的嵌合结构[4]。但TIR型转座子介导基因重复形成的机制仍不清楚,动物中此类基因重复也鲜有报道。
2021年7月13日,张勇研究组等在Nature Communications杂志上在线发表题为“DNA transposons mediate duplications via transposition-independent and -dependent mechanisms in metazoans”的研究论文。该研究首次大规模搜索了100个动物基因组,鉴定了TIR转座子介导的基因重复,并将其命名为pack-TIR。研究发现大部分基因重复的产生不依赖转座活性,符合缺口填补(gap-filling)模型;在黑腹果蝇群体基因组数据中找到的最年轻的多拷贝基因Ssk-FB4则符合新提出的模板跳转及转座(FoSTeST)模型。蛋白质组、自然选择压力分析及表型关联分析显示Ssk-FB4s编码功能蛋白。这些结果证明TIR转座子可以重塑基因结构,产生新基因;这是该研究组自发现动物中LTR转座子介导基因重复的成果以来又一项重大新发现,有助于填补领域空白。
在该研究中,研究团队首先收集了公共数据库UCSC中100个后生动物的基因组,计算分析鉴定出370个pack-TIR元件(图1A)。进而,该团队同时在黑腹果蝇群体中鉴定出一个多拷贝pack-TIR家族:Ssk-FB4(由FB4 转座子捕获基因Ssk所形成的新基因)。它们的基因结构和植物中pack-MULE相似,中间为捕获的外源序列,两侧为 TIR转座子序列(图1B)。
研究团队发现pack-TIR与TIR转座子数目成线性正相关(图2A),此外对灵长类谱系中pack-TIR的起源时间分析发现,它们零散分布在各演化枝上(图2B)。换言之,这些pack-TIR在各个时间段皆有起源,哪怕宿主编码的转座子已经丧失转座活性。因此可推断,大部分pack-TIR的产生不依赖于转座活性(transposition-independent)。多方面证据表明这些pack-TIR的形成符合缺口填补(gap-filling)模型(图2C):pack-TIR 的丰度与它们到母源序列的距离呈负相关,即TIR转座子倾向于捕获临近序列;67.6%事件含有微同源序列(pack-TIR与母源序列共享较短的相似序列);灵长类物种间的比较分析显示转座子和外源序列是先后插入的两次独立事件。随后研究人员发现果蝇中Ssk-FB4其形成过程与缺口填补模型不符,因此提出了复制叉停滞-模板跳转-转座(FoSTeST)这一新模型来解释多拷贝pack-TIR的产生(图2D)。该模型与缺口填补模型的不同之处在于,模板跳转后转座酶会立即将嵌合的pack-TIR转座到基因组其它位置。研究团队所采取的策略及发现对植物中pack-MULE的起源机制研究将具有启发意义。
除机制方面的深入分析,该研究团队也结合大量转录组数据和进化基因组数据探索了pack-TIRs的功能。一方面,团队发现pack-TIR更倾向于捕获外显子序列,且大部分与所在基因发生融合,具有转录信号(图3A)。另一方面,Ssk-FB4是一个罕见的编码功能蛋白的例子,所有拷贝不仅显示出了很高的转录和翻译水平(图3B-C),且各拷贝的非同义突变在细胞膜外结构域显著富集,暗示其受到正选择(图3D)。不仅如此,突变机制所赋予的Ssk-FB4嵌合结构使其更容易发挥新功能,有可能增加了果蝇抵御环境中致病微生物的能力(图3E-F);转座带来的拷贝数增加也使自然选择有更多的作用对象,推动了适应性进化更快发生。
该研究由中国科学院动物研究所、法国居里研究所、北京大学肿瘤医院等机构合作完成。动物所副研究员谭生军、博士后马慧静、博士后王金铂及肿瘤医院博士后王曼为本研究共同第一作者,动物所张勇研究员和居里研究所陈春龙研究员为共同通讯作者。王梦霞、尹浩东、张雅琼、张心莹、沈洁宇、王丹阳、Graham L. Banes、张治华、吴健民、黄勋、陈华、葛斯琴等合作者在文章写作、实验和计算分析等方面提供了大力支持。该研究得到了国家重点研发计划(2018YFC1406902, 2019YFA0802600)、国家自然科学基金(31701092, 31771410, 31970565, 91731302)、中国科学院青年创新促进会(2018112)等项目资助。
论文链接:https://doi.org/10.1038/s41467-021-24585-9
参考文献:
1. Esnault, C., J. Maestre, and T. Heidmann, Human LINE retrotransposons generate processed pseudogenes. Nature Genetics, 2000. 24(4): p. 363-367.
2. Tan, S., et al., LTR-mediated retroposition as a mechanism of RNA-based duplication in metazoans. Genome Research, 2016. 26(12): p. 1663-1675.
3. Grabundzija, I., et al., A Helitron transposon reconstructed from bats reveals a novel mechanism of genome shuffling in eukaryotes. Nature Communications, 2016. 7(1): p. 10716.
4. Jiang, N., et al., pack-MULE transposable elements mediate gene evolution in plants. Nature, 2004. 431(7008): p. 569-573.
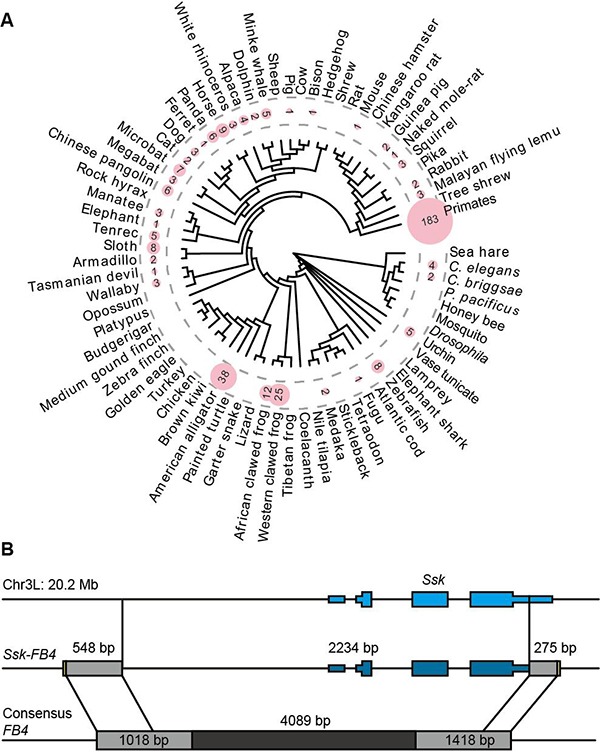
图1. 动物中pack-TIR分布及结构示意图。A)370个pack-TIR在100个动物中分布情况。圆圈内数字表示对应物种的pack-TIR数目。B)黑腹果蝇群体中Ssk-FB4的基因结构。蓝色片段为 Ssk的外显子,黑色和灰片段为 FB4序列。
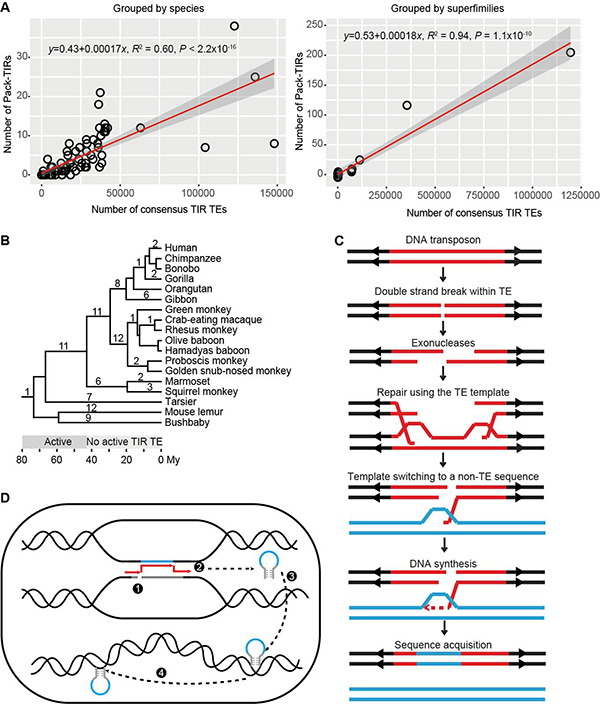
图2. 动物中pack-TIR形成的两种机制。A)各物种或各转座子超家族中pack-TIR与TIR转座子数目的线性关系。B)pack-TIR在灵长类动物演化树上的分布,数字表示pack-TIR出现在某一特定演化枝的个数,下方显示了转座子活跃与否的时间,单位为百万年。C)缺口填补模型示意图。红色片段及两侧箭头表示为DNA转座子,蓝色片段为外源序列。该图显示转座子内部双链断裂,也可能是转座子的自主切割引发缺口。D)FoSTeST模型示意图。1.复制叉停滞在转座子处并发生双链断裂;2.转座子和母源序列空间接近,导致修复时模板跳转产生嵌合片段;3.转座酶识别嵌合片段,切割插入到其它位置;4由于Ssk-FB4保留转座子的末端反向重复序列(TIR),在转座酶的帮助下增加其拷贝数。
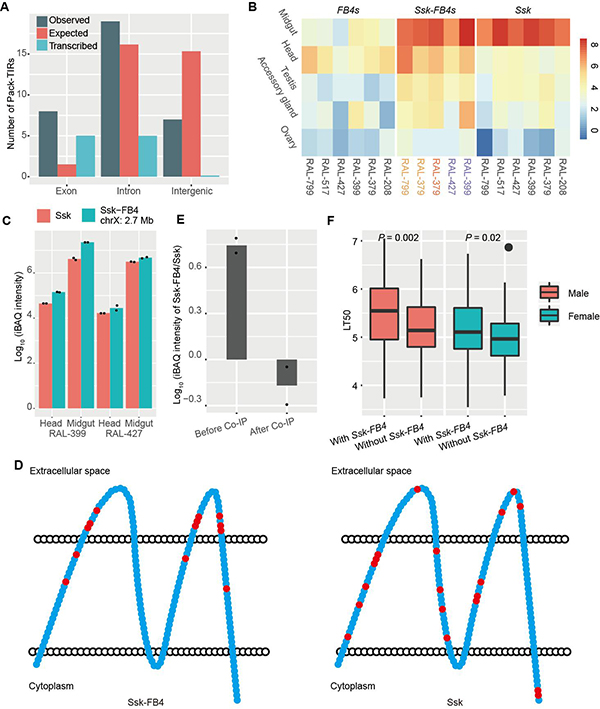
图3. pack-TIRs的表达、选择和关联分析。A)人类中pack-TIR捕获序列的类型分布情况。B)Ssk、Ssk-FB4、FB4在六个黑腹果蝇品系五个组织内的表达谱,表达值为log2(TpM值)。C)Ssk和Ssk-FB4蛋白水平的表达量。D)Ssk-FB4/Ssk蛋白非同义突变积累分布图。E)Ssk和Ssk-FB4蛋白与Mesh蛋白免疫共沉淀前后的蛋白定量数据。与母本基因Ssk不同,Ssk-FB4与Mesh互作水平较弱,暗示其具有新功能。F)黑腹果蝇DGRp品系在真菌Metarhizium anisopliae Ma549感染后的寿命半衰期值。






























