RNA修饰隐藏的神秘调控
2016年的最后一天,Nature Methods赶着发布了2016年度技术,不少人认为当年的年度技术一定是大火的CRISpR技术,然并卵,对于颇具前瞻性的年度技术盘点来说,一个并不常见的名词:Epitranscriptome analysis(表观转录组学)才是正解。
Epitranscriptome analysis这个名称是由希腊语“epi”作为前缀,指的就是除开已知功能或遗传性,任何添加到核苷酸上的修饰。几十年来,科学家们几乎都没有注意到RNA修饰,因为早在上个世纪60年代和70年代RNA上的标记就被发现了,但是大家只关注于tRNA和rRNA,以及DNA上的表观遗传修饰。
但随着科学家们发现了出现在所有RNA种类中的化学标记,动态添加或者去除这些标记的“Reader写手”和“Eraser橡皮擦”,重新点燃了对RNA修饰的兴趣。例如,从腺嘌呤上去除一个甲基基团的酶,与阿尔茨海默症患病风险之间的关联,表明了这种修饰在神经健康方面扮演了重要调节作用。
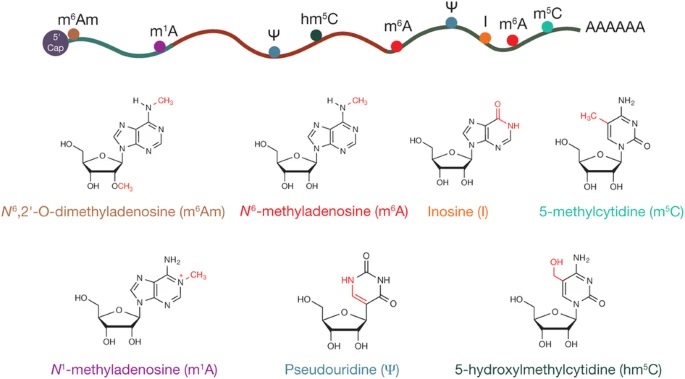
由此表观转录组学越来越受到科学家们的重视,成为了近来兴起的热门领域之一。迄今为止,在RNA上已发现了170多种化学修饰[1]。这些修饰大量分布在非编码RNA(ncRNA),特别是rRNA, tRNA和snRNA上,为ncRNA在翻译与剪接中发挥正常功能所必需。令人兴奋的是,研究人员发现m6A(N6-methyladenosine),m1A(N1-methyladenosine),m5C(5-methylcytidine),hm5C(5-hydroxylmethylcytidine),I(inosine)以及ψ(pseudouridine)等化学修饰也分布在真核生物mRNA上,影响mRNA的代谢与功能。特别是伴随着许多mRNA修饰酶(Writer)、去修饰酶(Eraser)和修饰识别蛋白(Reader)的新发现,mRNA化学修饰的可逆变化与动态调控重新激起了研究人员的兴趣。
图一来自[2]
表观转录组学研究范围
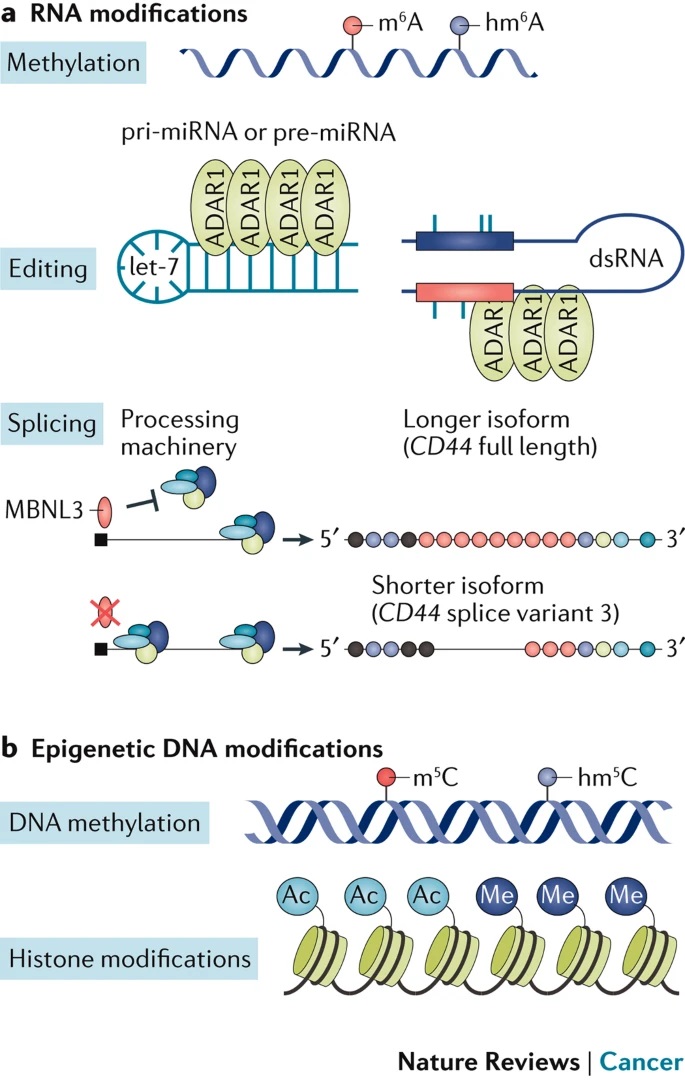
表观转录组学(epitranscriptomics,又称“RNA表观遗传学”)是指转录后 RNA 修饰,这些修饰给转录组带来了功能相关的变化。表观转录组修饰包括几个重要的 RNA 加工事件,包括 RNA 编辑、甲基化和剪接(图二)。
图二来自[4]
从是否编码蛋白质来说,RNA可以分为编码RNA(coding RNA)修饰和非编码RNA(non-coding RNA, ncRNA)两大类。前者就是指mRNA,后者则包括很多种类,如众所周知的tRNA和rRNA,参与RNA修饰的snoRNA等。
mRNA修饰研究最多的就是m6A 修饰,早在 20 世纪 70 年代,科学家们就在 RNA 中发现了 m6A 修饰,随后越来越多的研究证明m6A 修饰的重要性:m6A 修饰和 mRNA 的稳定性、剪接加工、翻译以及 microRNA 的加工有关;m6A 还和干细胞命运、生物节律相关,可以促使干细胞从自我更新状态转向细胞分化,研究人员发现,甲基化会缩短 mRNA 的半衰期,减少其丰度。可以说,m6A 修饰几乎影响 RNA 代谢的每个步骤。
而近年来,随着研究的深入,不少研究工作也从mRNA转为关注非编码RNA的甲基化对于疾病发生发展过程的重要作用,发现非编码RNA的m6A甲基化会在干细胞分化、癌细胞增殖等过程中起关键作用。同mRNA一样,lncRNA上也存在着多种化学修饰,m6A甲基化对于lncRNA来讲,可以调控lncRNA二级结构,lncRNA结合蛋白,以及lncRNA的ceRNA机制,靶基因的m6A修饰。
此外还有环状RNA上m6A修饰,这能影响circRNA和RNA结合蛋白(RBp)之间的相互作用,以及标记内源RNA,从而将其与外源RNA区分开,避免被自身免疫系统识别攻击。
Small RNA,例如microRNA,tRNA来源小RNA(tsRNA,包括tRF&tiRNA)等,其RNA分子上具有多种不同的修饰,这些修饰一方面能够调控small RNA的活性,另一方面也可以赋予它们新的功能。已知这些RNA修饰通过多种分子机制来发挥功能,如RNA修饰可以改变miRNA的靶向性或改变tsRNA(tRF&tiRNA)与RNA结合蛋白的亲和力,进而发挥生物活性等。Small RNA修饰谱分析是表观转录组学研究的新前沿,具有重要的科学意义和临床价值。
RNA 修饰领域最重要的成就
对于迄今为止RNA修饰领域最重要的成就,康奈尔大学Samie R. Jaffrey教授认为,“第一个用于绘制 m6A全转录组方法可能是表观转录组学领域中最重要的事件,现在已经被复制用于其他几种核苷酸修饰。 在全转录组作图之前,通过使用质谱法或其他分析测量检测水解 RNA 中修饰的核苷酸,发现修饰的核苷酸。这些方法是模棱两可的,尤其是对于低丰度的修饰:即使你有一个高纯度的 mRNA 制剂,你还是担心微量的污染转移 RNA (tRNA) 或核糖体 RNA (rRNA) 可能是修饰的来源。这使得很难确定修饰的核苷酸是否来自 mRNA。”
以色列特拉维夫大学医学院的Gideon Rechavi教授则表示,“在我看来,该领域的主要成就是通过mRNA 修饰揭示了一个新的、复杂的、高度敏感的、可调节的基因表达调控层。这一新的调控层利用了mRNA 的独特特性——即它是短暂的、高度结构化的、在细胞区室之间移动并通过转录放大的。这些影响部分是由“阅读器”介导的,例如甲基特异性结合蛋白,它的鉴定是修饰领域的一个里程碑。基因表达的调节也通过“写手”和“橡皮擦”的修改安装和删除之间的相互作用进行调整。在过去的十年中,出现了几个重要的教训。首先,mRNA 修饰非常普遍,数千个基因转录本被修饰。有趣的是,一些修改聚集在特定的转录位置;例如,肌苷主要存在于重复的 Alu 序列中,m6A 优先修饰终止密码子附近和内部外显子、 AUG 起始密码子周围的 m1A 簇,这表明每个修饰通过不同的模式起作用行动。此外,一些修饰,如 m6A 和 m1A,在人和小鼠之间表现出高度的保守性。
另一个重要的成就是发现特定的修改可以通过不同的行为模式,通过不同的读取,依赖于上下游。还有一个重要发现是一些 mRNA 修饰的动态特性,可以对环境刺激做出快速反应; m6A 和 m1A 已经证明了这种动态特性。 mRNA 修饰的核心作用体现在异常修饰对人类和小鼠早期发育以及人类癌症、炎症和神经变性的破坏性影响,进一步强调了这一调节层的重要性。”
东京大学的Tsutomu Suzuki教授表示,“以前关于 RNA 修饰的生化研究主要集中在经典的非编码 RNA,包括 tRNA、rRNA 和小核 RNA (snRNA),因为这些RNA 在细胞中含量丰富。然而,最近,使用 NGS 技术对 RNA修饰进行全转录组分析已定出几种碱基修饰,包括 mRNA 中的肌苷 (I)、m6A、m5C、Ψ 和 m1A 以及长链非编码 RNA。这反过来这大大拓宽了表观转录组的概念。
在过去的十几年中,通过反向遗传学和质谱法,科学家们识别出了埋藏在模式生物基因组中的RNA 修饰酶。使用这种方法,我们成功鉴定了 40 个RNA 修饰基因。
还值得一提的是,与疾病相关的外显子组测序有助于解析RNA 修饰酶突变如何引发了许多人类疾病。”[5]
如何研究RNA修饰
从目前的研究成果来看,表观转录组影响深远,是作为将可塑性叠加在其他基因组上连线转录组上的一种手段。表观转录组“代码”不仅可以启用或增强 RNA 催化或 RNA 依赖性反应中的特定化学反应,还可以改变 RNA 结构-功能关系,从而以时空和信号依赖性方式提供额外的基因调控层。
然而,表观转录组的功能研究落后于表观基因组的功能研究,这是因为缺乏可以在转录组范围内检测这些表观转录组标记的灵敏且稳健的技术。研究表观转录组存在几个主要挑战。首先,大多数 RNA 修饰不能通过高通量测序直接检测到。因为对 RNA 的化学修饰通常不会改变修饰碱基的碱基配对特性,逆转录 (RT) 将简单地擦除这些修饰,并使它们与常规 RNA 碱基无法区分。其次,虽然 rRNA、tRNA 和 snRNA 很丰富,但其他类型的RNA,例如 mRNA 和长链非编码 RNA (lncRNA),丰度可能较低。第三,缺乏现有的计算工具来促进从测序数据识别修饰位点的能力。
幸运的是,近年来,针对不同表观转录组的研究方法取得了重大进展。这些新工具帮助研究人员确定 RNA 修饰的位置,并揭示这些修饰在整个转录组中的不同分布模式。当这些方法与其他新兴工具(例如基因组编辑工具)结合使用时,RNA 修饰酶的靶标就已确定。此外,这些技术还揭示了不同生理条件下不同表观转录组标记的动态性质。同时这些工具使人们能够发现选择性识别特定表观转录组标记并确定其功能的“阅读器”蛋白。因此,新的研究工具不仅可以对表观转录组进行全面分析,而且还是功能表观转录组研究的宝贵资源 [2]。
⑴ 检测分析方法
研究 RNA 修饰的方法包括是液相色谱(LC-MS) 、基于高通量测序的方法和芯片分析等方法。
① mRNA&lncRNA表观转录组芯片
RNA修饰的潜在功能不仅取决于其所修饰的是何种基因转录本,同时也取决于被修饰部分在该转录本中所占的百分比。然而,目前大部分转录组水平的的RNA修饰检测方法着重于寻找转录本上的修饰位点,不能够定量地检测被修饰转录本的百分比。这一类定量信息的缺乏已引起越来越多科研工作者的关注。
mRNA修饰的潜在影响既取决于其分子效应,也取决于被修饰转录本的百分比。例如,一种可以加速mRNA降解的修饰,如果只有1%的转录本被修饰,显然不太可能产生任何生物功能,然而当一种修饰可以促使mRNA翻译成新的蛋白亚型时,即使修饰水平很低,也可能产生重要的生物功能。当前的m6A和Ψ检测方法局限性在于缺乏修饰程度的定量信息。m6A的调控作用可以通过pulldown m6A的方法,检测特定序列在不同状态下的相对富集程度进行推断,但不能从这些数据中获得被修饰mRNA的绝对量。新的可定量m6A和Ψ的高通量检测方法,将极大的促进该领域的发展[7]。
另一个重要的问题是阐明RNA修饰的化学计量的动态变化。目前,表观转录组学研究的重点大多是哪些位点被修饰,而不是RNA中每个被修饰位点的占比。低通量分析mRNA和病毒RNA的m6A修饰位点显示,任何m6A修饰位点的占比都不会达到100%。修饰的化学计量变化可能是一种RNA生物学修饰的动态变化参数。修饰可以影响mRNA的结构,和(或)对RBps的招募。任何特殊位点的修饰,会导致同一mRNA群体仅仅由于结构或是结合reader的不同,分为两个mRNA亚群。因此,改变修饰的化学计量数,可能是同一个RNA转录本行使不同功能的一种机制。目前急需可以检测修饰化学计量数的高通量方法,以阐明表观转录组学在这一方面的问题[8]。
Arraystar mRNA&lncRNA表观观转录组芯片结合了双荧光通道芯片技术与RNA修饰免疫共沉淀技术,在转录本水平对RNA修饰进行定量检测。定量的表观转录组图谱可为RNA修饰调控研究提供重要信息。
此外,还有Arraystar small RNA修饰芯片,康成提供的相关技术服务在单张芯片可定量miRNA,pre-miRNA和tRNA衍生的small RNA(tsRNA,包括tRF&tiRNA)的碱基修饰。可检测的修饰包括:8-氧代鸟嘌呤(o8G),7-甲基鸟苷(m7G),N6-甲基腺苷(m6A),假尿苷(Ψ)或5-甲基胞苷(m5C)。
索取康成生物Arraystar 表观转录组芯片
②液相色谱-质谱法
LC-MS 是一种强大的技术,能以出色的灵敏度和高特异性检测修饰过的RNA核苷。然而,在测量 mRNA 修饰的背景下,有两个关键限制。
首先,LC-MS 进行位点特定检测的能力有限。在 mRNA 的背景下,迄今为止,MS 仅适用于完全消化的核苷,因此能够估计样品中修饰的“整体”水平,但排除了将该修饰分配到单个位点的可能性。当应用于部分消化的 RNA 寡核苷酸时,LC-MS 原则上还可以提供位点特定信息。然而,这种分析通常需要数十到数百纳克的至少部分纯化的分子,这对于 mRNA 来说是不现实的,因为它具有高度异质性和低丰度 的特性。
其次,与 tRNA 和 rRNA 相比,mRNA 中修饰的相对水平越低,因此解释 LC-MS 结果变得越来越困难。即使来自高度表达的 tRNA 和 rRNA 的低水平污染(永远无法完全避免,尽管它们可以在一定程度上基于 tRNA 特异性或 rRNA 特异性核苷进行估计),也可能导致对mRNA修饰水平的高估。m6A 是 MS 广泛进行的第一个mRNA 修饰分析,在这方面施加的限制较少,因为它在 mRNA 中高度丰富,但在 tRNA 中不存在,并且仅存在于 rRNA 中的两个位点(因此,污染可能导致低估实际 m6A 水平)。
相比之下,绝大多数非 m6A 表观转录组在 mRNA 中都非常罕见,在 tRNA 和 rRNA1 中更为普遍,这严重限制了 LC-MS 了解其丰度和动态的能力。在一些情况下,通过 LC-MS 估计的修饰丰度(基于该修饰被认为在 mRNA 中以高水平存在)和基于基因组方法(通常无法检测 mRNA 中的大量修饰);因此,这些差异的根源可能在于 mRNA 部分中的 tRNA 或 rRNA 污染物。另一种可能性是某些修饰是 RNA 损伤的结果,这可能导致核苷酸的烷基化和氧化。这种随机散布在整个转录组中的非酶促修饰可以通过 LC-MS 检测到,但不能通过基因组方法检测到,这需要在特定位点积累信号。
③基于测序的方法
在过去十年中,基于高通量测序分析各种修饰已经成为了该领域进步的关键驱动力。这些方法可以告知 mRNA 中修饰的存在及其在转录组中的精确位置。
与 LC-MS 相比,原则上单个工作流程就可以获知所有修饰的丰度,而使用基因组方法识别修饰需要为每个修饰开发专用且独特的工作流程。这种方法需要克服的根本挑战是,大多数修饰在标准测序中是“不可见的”,也就是说,它们在将 RNA 逆转录为 cDNA 后不会留下任何痕迹,这是基于 Illumina 的标准测序的先决步骤方法。因此,科学家们已经开发了一套方法来呈现不同的可见修改。对于 m6A,主要检测方法依赖于抗 m6A 抗体,这些抗体用于选择性免疫沉淀含有 m6A 的短 RNA 片段。
尽管免疫沉淀是一种通用方法,原则上可以用于检测任何修饰,但实际上,基于抗体的方法在绘制非 m6A 表观转录组的图谱方面的效用相当有限。曾经研究人员尝试使用它来绘制 m1A 或 ac4C36 的图谱,现在被认为会导致大量假阳性位点,这可能是由于抗体交叉反应造成的。抗 m6A 抗体也存在这种交叉反应性;然而,鉴于 m6A 的丰度相对较高,信噪比仍然可控。相比之下,以较低数量级的丰度存在的修饰导致非特异性与特异性结合事件的比率急剧增加。
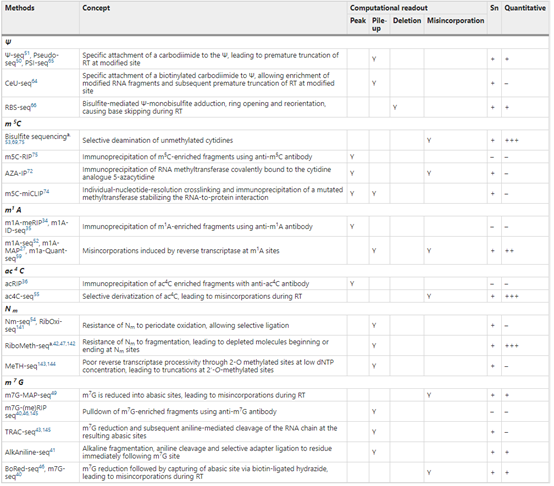
因此,对于绝大多数非 m6A 表观转录组,已经开发出利用修饰碱基的独特化学性质使它们在逆转录后可见的方法。这是通过不同的策略实现的,例如,通过改变修饰碱基身份的修饰特异性化学物质或通过将大量残基特定连接到修饰碱基上导致逆转录过程中过早截断(见下表)。这些实验方案通常会导致修改位点的错配或缺失,或者导致读取的“堆积”,这些读取选择性地在特定位置开始或结束,然后可以从中推断出修改的存在。此类化学基因组学方法的主要优势在于其出色的、通常为单核苷酸的分辨率,以及它们能够提供修饰水平的相对定量,有时甚至是绝对定量[1]。
比如康成生物建立了一种高灵敏的绝对定量的实时荧光定量pCR定量分析方法,可以在单核苷酸分辨率水平上准确检测RNA中m6A修饰位点, 并对未知模板进行定量分析。该方法可成功地应用于实际生物样品中m6A修饰的精确检测,即使是低丰度RNA的样本。
索取康成Arraystar m6A单碱基分辨率芯片技术资料>>
近年来也有一些新的测序技术陆续出现,比如2017 年,北京大学伊成器课题组和以色列魏茨曼研究所的研究团队分别独立报道了单碱基分辨率水平检测 m1A 的新方法——m1A-MAp 和m1A-Seq。2019 年,芝加哥大学 Bryan C. Dickinson 团队与何川团队合作,研究开发了可快速选择逆转录酶的进化平台,并利用该平台研究 mRNA 上的 m1A 修饰,实现了单碱基水平 m1A 的检测。
另外,直接的RNA纳米孔测序也备受关注,RNA表观遗传学的先驱人物、康奈尔大学的Christopher Mason表示,“过去,我们通常是利用抗体或化学分析来推断RNA的修饰状态。直到最近,我们才开始进行直接的RNA测序。利用纳米孔测序仪,我们能够直接测序RNA,而不需要逆转录。我们第一次直接测定RNA修饰:整个分子存在哪些修饰,又存在哪些异构体。”
所以可以说,纳米孔测序能完整测序,读出RNA序列的m6A,m5C修饰(一般是读取单分子序列时比无修饰碱基有延迟),读长优势使之对可变剪切研究和异构体更有优势,不用拼接,也不容易丢失罕见分子,但是就是费用比较贵。
在最近的一篇热点文章“The Architecture of SARS-CoV-2 Transcriptome SARS-CoV-2”中,研究人员就用MinION纳米孔测序仪进行了DRS测序,获得了879,679个reads(1.9 Gb),结果不仅证明了病毒转录本占据主导地位,而且纳米孔DRS基于RNA的单分子检测,提供了独特的机会来检查单个RNA分子的多个转录组特征,解析了新冠病毒RNA修饰特点[9]。
⑵目前已知的RNA表观转录修饰:
m6A
m6A是真核生物mRNA上含量最丰富的化学修饰,由甲基转移酶复合物(包含METTL3,METTL14,WTAp,KIAA1429,RBM15,RBM15B)催化产生,可被去甲基化酶ALKBH5或FTO去除。目前已发现了多种特异性识别m6A位点的蛋白或复合物,包括YTH家族蛋白(YTHDF1-3,YTHDC1)、转录起始复合物eIF3、核糖核蛋白(HNRNpA2B1,HNRNpC)以及RNA结合蛋白SRSF2。m6A主要分布在终止密码子附近和3’UTR区,影响RNA配对、改变RNA二级结构、或被蛋白直接识别,进而调控mRNA的成熟、可变剪接、稳定性和翻译过程。与A-U配对相比,m6A-U配对较不稳定,引发RNA内部双链解旋与二级结构转变。m6A常在双链与单链的过渡区域堆叠,增强RNA转变后构象的稳定性。去甲基化则可以使mRNA恢复原来构象。这种构象转变可能导致mRNA与不同蛋白的相互作用改变,从而产生不同的生物学效应。m6A能直接被特定蛋白的疏水结构域所识别。比如,YTH家族蛋白可以特异性的识别m6A,特别是GGm6ACU保守序列。其成员之一YTHDC1识别并结合m6A,调控靶向mRNA的可变剪接。而另一成员YRHDF2与m6A结合后,招募CCR4-NOT复合物,促进靶向RNA的降解。在UV辐射或热休克反应中,转录起始复合物eIF3与5’UTR区的m6A结合,促进帽非依赖的翻译过程。编码区的m6A可被SRSF2识别,参与脂肪生成的调控。在果蝇中,YTHDC1同源蛋白识别性别致死mRNA上的m6A位点,调控其可变剪接,从而控制果蝇的性别。如前所述,m6A介导的mRNA稳定性调节也对干细胞分化与生物节律时钟控制十分重要。此外,m6A也能通过影响mRNA与tRNA反密码子的配对速率与保真度,从而影响翻译延伸。
研究方法方面,Arraystar m6A单碱基分辨率芯片利用RNA酶MazF对m6A敏感的特性,可在单碱基分辨率水平定位m6A位点,并对其进行定量。该芯片具有其它现存技术难以企及的优势,是研究m6A动态变化、生物学功能以及疾病相关性的强大工具。
m1A
m1A是新近发现的可逆的表观转录修饰,能被RNA修复酶ALKBH3去除,目前尚未发现明确的m1A修饰酶与修饰识别蛋白。与m6A不同,m1A的表达丰度较低,主要分布在mRNA的5’UTR区,可能参与调节翻译起始过程。m1A能完全阻止Watson-Crick配对,引起RNA双链解旋,并促进RNA-蛋白的静电相互作用或RNA可变二级结构的形成。m1A的生物学功能仍属未知。有研究发现,在热休克或营养匮乏等压力条件下,细胞内的m1A表达水平上升,可能是通过促进帽依赖性翻译,参与细胞的应激反应。
m5C与hm5C
m5C广泛分布于tRNA与rRNA上,具有稳定tRNA二级结构、影响反密码子环构象、维持rRNA翻译保真等功能。新近的RNA测序结果发现,mRNA的编码区与非编码区上存在8000多个m5C位点,而且相当一部分位点集中在5’UTR与3’UTR区。m5C可由甲基转移酶NSUN2或TRDMT1催化形成,被双加氧酶TET氧化形成hm5C。hm5C可能经过进一步氧化形成f5C,进而变回胞嘧啶核苷(C)。m5C不影响碱基配对,但可能增强碱基堆叠以及RNA与蛋白的疏水作用。m5C具有多种生物学功能。p16 mRNA在被NSUN2酶添加m5C修饰后,其降解被抑制,稳定性增强。在细胞周期中,NSUN2的表达受到精密调控。NSUN2可在CDK1 3’UTR区添加m5C修饰,促进其翻译;同时在CDKN1B的5’UTR区添加m5C修饰,抑制CDKN1B的翻译。二者共同作用,增强细胞的增殖能力。m5C还与衰老相关基因的翻译控制有关,过表达NSUN2可以延缓复制性衰老的发生。作为m5C的氧化产物,hm5C也能增强翻译效率。hm5C在果蝇的脑中表达量很高,可能参与果蝇的脑部发育。
pseudouridine (ψ)
假尿嘧啶核苷ψ,常被成为第五类核苷酸,由尿嘧啶核苷(U)异构化形成。在人的细胞和小鼠组织的mRNA中,ψ/U的比率约为0.2-0.6%。尿嘧啶与假尿嘧啶的异构化反应由pUS酶单独,或与H/ACA核糖核蛋白一起,催化完成。假尿嘧啶能减少RNA构象的可变性,增强碱基配对稳定性以及与蛋白之间的的极性相互作用。假尿嘧啶可能调控mRNA稳定性和基因表达,参与酵母的热休克反应,但具体机制目前尚不明确。
Inosine (I)
肌苷修饰,常称为A-to-I编辑,是高等真核生物里最常见的一种RNA编辑方式,由腺苷酸脱氨酶ADAR完成。A-to-I编辑主要发生在非编码区或内含子区的Alu元素内。A-to-I编辑完全改变了碱基配对特性,AU配对转变为IC配对,从而改变所编码的氨基酸。比如,A-to-I编辑将大脑谷氨酸受体中的谷氨酰胺重编码为精氨酸,导致了钙离子通透性的改变。此外,A-to-I编辑还具有改变可变剪接、调节miRNA的产生与功能、以及监控先天性免疫反应等作用。
ac4C
2018年,美国NIH的Shalini Oberdoerffer教授在Cell发表研究论文:Acetylation of Cytidine in mRNA promotes Translation Efficiency,首次揭示mRNA上存在大量ac4C修饰,并且ac4C影响mRNA的稳定性与翻译效率。
N4-acetylcytidine (ac4C),N4位乙酰胞嘧啶,是真核原核生物中保守的化学修饰,早期研究认为ac4C主要存在tRNA和18S rRNA上。而近期研究显示,mRNA上也存在大量的ac4C,其丰度甚至不低于mRNA携带的m7G帽子修饰。NAT10是目前鉴定的唯一同时具有乙酰化酶结构域和RNA结合结构域的蛋白,因此被认为是RNA ac4C修饰酶。
由此,乙酰化作为一类新的 mRNA 修饰,将表观转录组扩展到已知类别的甲基化和异构化之外。这些工作还扩展了 mRNA 修饰的已知库,并确定了多种化学修饰的存在,mRNA 修饰的新模式在未来很可能会继续出现。进一步了解这些修饰的功能、调节它们在特定转录本上的存在的途径和机制,以及它们在各种生理和疾病背景下的功能,将有助于更全面地了解表观转录组在基因表达中的作用[6]。
TGM修饰
5’端超甲基化的TMG修饰是最早被发现的修饰之一。在许多被RNA聚合酶II转录的RNA中,尤其是非编码RNA,例如剪接体的重要组成成分snRNA,5’端的m7G甲基帽子会被超甲基化,形成具有三个甲基的N2, N2, 7-三甲基鸟苷(TMG)帽子,广泛存在于真核生物中。三甲基鸟苷合成酶(Tgs1)在许多生物体中已经被证实是合成TMG帽子的唯一甲基酶,在进化上从酵母到人类都很保守。由于体外实验的局限性,TMG修饰对剪接体RNA的调控机制还存在很多不清楚的地方,并且在大多数多细胞模式生物中都无法直接从体内对TMG修饰进行研究,因此,TMG修饰已经被发现了五十多年,但是其在多细胞生物中的功能并不明确。
(生物通:万纹)
参考文献:
1. The epitranscriptome beyond m6A,Nature Reviews Genetics volume 22, pages119–131 (2021)
2. Epitranscriptome sequencing technologies: decoding RNA modifications,Nature Methods volume 14, pages23–31 (2017)
3. Detecting RNA modifications in the epitranscriptome: predict and validate,Nature Reviews Genetics volume 18, pages275–291 (2017)
4. RNA editing-dependent epitranscriptome diversity in cancer stem cells,Nature Reviews Cancer volume 17, pages381–392 (2017)
5. RNA modifications: what have we learned and where are we headed? Nature Reviews Genetics volume 17, pages365–372 (2016)
6. mRNA acetylation: a new addition to the epitranscriptome,p. Cody He & Chuan He Cell Research volume 29, pages91–92 (2019)
7. Wendy V. Gilbert, Tristan A. Bell, Cassandra Schaening. Science (2016)
8. Cole J.T. Lewis, Tao pan, Auinash Kalsotra. Nat Rev Mol Cell Biol (2017)
9. The Architecture of SARS-CoV-2 Transcriptome,Cell,April 23,2020