Kenichi Tsuda教授团队揭示十字花科植物免疫进化机制
南湖新闻网讯(通讯员 韩晓伟)近日,我校农业微生物学国家重点实验室和植物科学技术学院Kenichi Tsuda教授团队的最新研究成果发表,研究揭示了十字花科植物pTI免疫反应的进化机制。
在植物长期进化过程中,胁迫响应基因的变异对于植物适应环境变化至关重要。但是,植物在受到胁迫时基因转录的进化机制尚不清楚。植物进化出膜定位的模式识别受体(pRR)识别病原微生物表面保守的微生物相关分子模式(MAMps),激活模式诱发的免疫反应(pTI)。例如,细菌鞭毛蛋白N端的寡肽flg22可以被拟南芥受体FLS2识别,引发包括MApK磷酸化、转录重编程和激素合成等一系列pTI免疫反应,从而提高植物对病原菌的抗性。目前人们对pTI进化机制的了解仅仅局限于pRR的进化,而对于不同植物间pTI免疫反应的保守性和进化机制还知之甚少。
研究人员用flg22处理六个拟南芥品系以及三种十字花科近缘种(包括荠菜Capsella rubella、碎米荠Cardamine hirsuta和盐芥Eutrema salsugineum),发现四种十字花科植物均能感应flg22并引发pTI早期反应,但是flg22诱发pTI反应导致的植物生长抑制和病原菌抗性在四种植物间却存在明显差异。
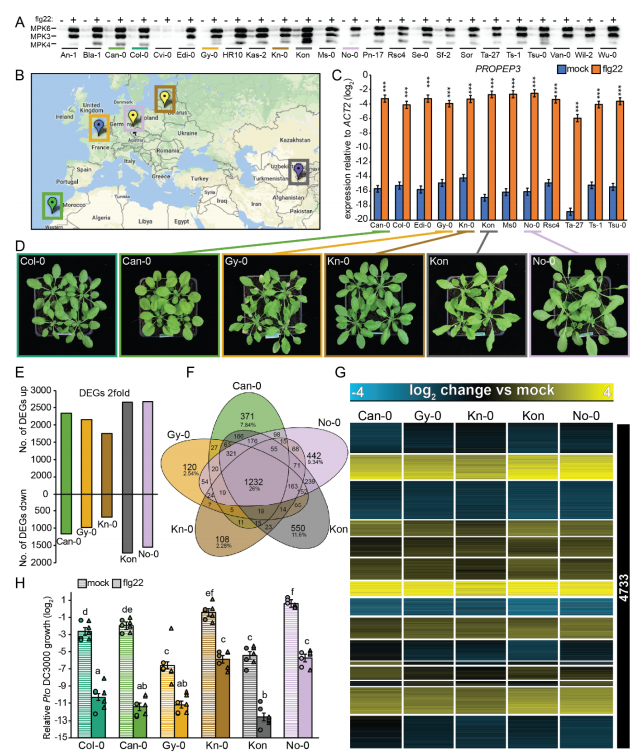
不同遗传背景和地理来源迥异的拟南芥品系对flg22的转录响应高度一致
为研究pTI免疫反应在转录水平的进化,研究人员比较分析了六个拟南芥品系以及三种十字花科植物受flg22处理后的转录组,发现四种十字花科植物既共有一些差异表达基因(DEG),也有相当多的DEG呈现物种特异性;然而六个遗传背景和地理来源迥异的拟南芥品系对flg22的响应却高度保守。但是十字花科物种间对flg22响应的差异与其系统发育却不一致。研究人员进一步发现WRKY转录因子结合位点在物种特异型flg22响应基因的5’-顺式调节区域富集,可能驱使物种特异性基因转录的进化。同时,研究还发现编码区序列差异与转录组差异无相关性,而纯化选择可能维持十字花科植物共有flg22响应基因5’-调节区域的保守性。此外,研究结果还表明不同十字花科植物在flg22处理后的代谢组具有物种特异性。
研究者这样说,这个研究的发现揭示了十字花科植物pTI过程中基因转录的进化机制,为进一步了解植物在生物胁迫下转录水平进化机制提供了线索。
审核人:津田贤一
【英文摘要】
plants recognize surrounding microbes by sensing microbe-associated molecular patterns (MAMps) to activate pattern-triggered immunity (pTI). Despite their significance for microbial control, the evolution of pTI responses remains largely uncharacterized. Here, by employing comparative transcriptomics of six Arabidopsis thaliana accessions and three additional Brassicaceae species to investigate pTI responses, we identified a set of genes that commonly respond to the MAMp flg22 and genes that exhibit species-specific expression signatures. Variation in flg22-triggered transcriptome responses across Brassicaceae species was incongruent with their phylogeny, while expression changes were strongly conserved within A. thaliana. We found the enrichment of WRKY transcription factor binding sites in the 5’-regulatory regions of conserved and species-specific responsive genes, linking the emergence of WRKY-binding sites with the evolution of gene expression patterns during pTI. Our findings advance our understanding of the evolution of the transcriptome during biotic stress.
论文链接:https://academic.oup.com/plcell/advance-article/doi/10.1093/plcell/koab073/6159620?searchresult=1






























