朱正江课题组发表基于离子淌度质谱的多维代谢物鉴定技术和数据库
中国科学院上海有机化学研究所生物与化学交叉研究中心朱正江研究员课题组在Nature Communications杂志在线发表了题为“Ion Mobility Collision Cross-Section Atlas for Known and Unknown Metabolite Annotation in Untargeted Metabolomics”的研究论文(https://www.nature.com/articles/s41467-020-18171-8)。该工作发展了基于离子淌度质谱的碰撞截面积数据库(AllCCS),并开发了基于离子淌度质谱的多维代谢物分析技术用于生命体内已知和未知代谢物的化学结构鉴定。朱正江课题组博士生周智伟是论文的第一作者,中国科学院上海有机化学研究所生物与化学交叉研究中心为第一单位。
离子淌度质谱(Ion Mobility-Mass Spectrometry)是一类能够根据分析物离子的尺寸、形状和电荷进行气相分离和检测的质谱技术,一次分析能够同时提供多个维度信息,包括精确质量数(MS1)、二级碎片谱图(MS/MS)和碰撞截面积(CCS)数据等,有效提升对复杂生物样品代谢组的定性和定量分析能力。朱正江课题组一直致力于发展基于离子淌度质谱的代谢组学和脂质组学技术。2016年,朱正江研究员课题组首次发展了机器学习算法来计算代谢物的碰撞截面积的方法(Anal. Chem. 2016, 88, 22),开发了大规模碰撞截面积数据库MetCCS应用于代谢组学研究 (Bioinformatics 2017, 33, 14; Curr. Opin. Chem. Biol. 2018, 42)。针对脂质类化合物的结构特点,发展了基于脂质的碰撞截面积预测方法LipidCCS(Anal. Chem. 2017, 89, 17)和四维脂质鉴定鉴定技术LipidIMMS应用于非靶向脂质组学研究(Bioinformatics 2019, 35, 4; Trends Anal. Chem. 2019, 116)。
在上述工作的基础上,课题组再次发展了一个全新的碰撞截面积预测方法和数据库AllCCS, 具有以下特点:(1)提供了一个世界上最大、最全面的小分子碰撞截面积数据库平台,包含5000多个实验碰撞截面积和1100多万个预测碰撞截面积;(2)发展了新一代的碰撞截面积预测算法,能将预测准确性提升至2%以内,并广泛适用于不同化学结构的小分子化合物;(3)发展了特征结构相似性得分(RSS),实现了对碰撞截面积预测误差的估计;(4)开发了基于离子淌度质谱的多维代谢物分析技术,提升已知代谢物鉴定的准确性,并能用于未知代谢物鉴定,为未知代谢物的鉴定提供了一种全新的方法。
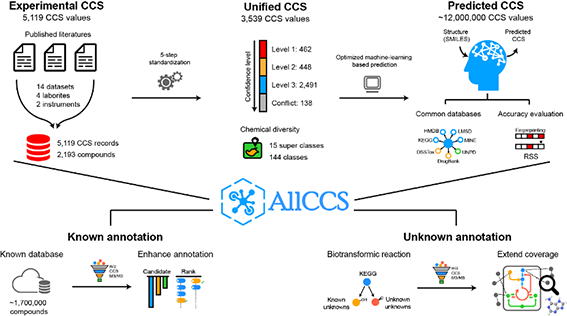
基于离子淌度质谱的多维代谢物鉴定和碰撞截面积数据库AllCCS
复杂生物样本中的代谢物鉴定是代谢组学研究中面临的最大挑战。目前,代谢物鉴定主要依赖于二级碎片谱图(MS/MS),其鉴定范围受限于标准谱图的数目,难以注释未知代谢物。而传统的小分子化合物库如pubChem,尽管含有多达1亿种小分子,但是大多是非天然化合物,结构冗余度高,难以用于发现生物内源性的新代谢物。针对未知代谢物鉴定的挑战,朱正江课题组进一步开发了基于离子淌度质谱的多维代谢物鉴定技术。该方法首先基于生命体内普遍存在的178个生物化学反应,从已知代谢物构建新的10万种未知代谢物化学结构。 利用AllCCS及其他MS/MS工具进行多维质谱信息的预测,并通过多维匹配和打分来提升代谢物鉴定的准确性。该技术能够有效地去除假阳性注释,提升了超过80%代谢物的定性分析准确性,进而实现了大规模检测和鉴定未知代谢物。
为方便用户使用,AllCCS平台(http://allccs.zhulab.cn/)已在阿里云服务器上免费向学术用户开放。进一步的商业用途需要联系朱正江研究员进行授权使用。AllCCS算法已经申请了国家发明专利。AllCCS软件申请了国家软件著作权的保护。
该工作得到了国家重点研发专项、国家自然科学基金委、中国科学院、上海市科委等的大力资助。
文章链接:https://www.nature.com/articles/s41467-020-18171-8






























